Indice
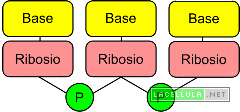
Il termine DNA è l’acronimo di Acido Deossiribo Nucleico; strutturalmente il DNA è una lunga molecola formata da basi azotate, zuccheri ribosio e ponti fosforici. In alcuni organismi, tra cui i batteri, il DNA è circolare e pertanto non possiede estremità libere mentre in altri organismi, perlopiù eucariotici, la molecola è lineare, spiralizzata o diversamente organizzata in cromosomi. Se consideriamo i batteri, che sono procarioti, e gli eucarioti possiamo osservare che la localizzazione del DNA differisce dal punto di vista spaziale. I batteri non possiedono il nucleo perciò il DNA è in soluzione con il liquido citoplasmatico mentre gli eucarioti, che invece presentano il nucleo, possiedono il DNA, più o meno spiralizzato dentro la membrana nucleare. Un discorso a parte è da fare per i virus la cui informazione genetica è conservata sia nel DNA che nell’RNA; ad ogni modo i virus non possiedono nucleo ma soltanto un rivestimento esterno di natura proteica. Non esistono grandi differenze nella struttura del DNA tra procarioti ed eucarioti ad esclusione della dimensione, espressa in numero di basi per singolo filamento, e dai processi di utilizzo che spesso operano in modo molto diverso.
Con le conoscenze scientifiche attuali è quasi superfluo definire il DNA come il materiale genetico caratteristico delle cellule e dei virus. Eppure la certezza di questa affermazione è stata garantita soltanto da pochi decenni; prima del superbo lavoro di Watson e Crick, e del loro team, mancavano le idee e gli strumenti per poter capire l’intimo funzionamento della cellula dal punto di vista della genetica e della trasmissione dei caratteri ereditari.
I primi esperimenti che indagarono circa la capacità di contenimento dell’informazione genetica all’interno del DNA risalgono alla prima metà del XX secolo; in quel periodo si comprese che il batterio Pneumococcus è capace di uccidere i topi. Successivamente si osservò che questo batterio possiede una molecola adesa alla sua membrana cellulare che ne determina la virulenza. La presenza della molecola, appartenente alla classe dei polisaccaridi, determina anche un aspetto morfologico della superficie stessa: l’espressione della proteina determina un batterio di tipo liscio o “S” (smooth) mentre l’assenza determina un tipo ruvido “R” (rough). Il tipo liscio è capace di uccidere il topo mentre il tipo ruvido non lo è.
Trattando il tipo liscio con il calore e iniettando il risultato del processo in un topo si notò che questo non moriva. Il calore aveva distrutto il batterio che era incapace di infettare i tessuti dell’ospite. Se, però, si iniettava una soluzione contenente il batterio liscio ucciso dal calore ed il batterio ruvido, che è incapace di uccidere, il topo moriva proprio come se fosse stato infettato con il tipo virulento. Questo dimostrava che c’era un principio trasformante capace di modificare un tipo di batterio non letale in un tipo letale. L’elemento fu subito purificato e si notò che era un acido deossiribonucleico.
Per dimostrare che il DNA è anche il materiale genetico dei virus (più avanti vedremo che c’è anche l’RNA) si utilizzò il fago T2. In linea generale il fago, o batteriofago, è un virus che infetta i batteri portando alla loro distruzione mediante lisi della membrana. Il fago T2 infetta Escherichia coli e la sua azione distruttiva avviene quasi immediatamente. Dopo circa mezz’ora dall’inserimento di una relativamente bassa quantità di T2 in una coltura di E. coli si osserva che il batterio lisa e rilascia la progenie di T2 che, nel frattempo, si è sviluppata.
Marcando preventivamente l’acido nucleico dei fagi T2 con uno fosforo radioattivo e utilizzando un isotopo radioattivo dello zolfo per le proteine si osservò che, a seguito dell’inoculazione in coltura di E. coli, gli involucri virali separati dai batteri avevano ricevuto circa l’80% dello zolfo marcato. Una simile quantità di fosforo radioattivo fu rivelata nella progenie e, pertanto, si dimostrava che ambedue gli elementi instabili erano stati incorporati sia nel rivestimento proteico sia nel DNA. Dopo la prima divisione lo zolfo radioattivo era presente in tracce mentre il fosforo radioattivo era valutabile in un quantitativo pari a circa il 30%. Mediante questo esperimento fu possibile determinare che anche il DNA è la molecola adibita alle funzioni di memorizzazione dell’informazione genetica.
Il DNA è anche il materiale genetico delle cellule animali. Per poter verificare questa affermazione si inoculò del materiale genetico estraneo all’interno di alcune cellule animali in coltura. I frammenti di DNA erano responsabili della sintesi dell’enzima timidina chinasi e si osservò che, dopo l’immissione del frammento, le cellule acquisivano la capacità di sintetizzare l’enzima in questione. La dimostrazione che il DNA è adibito al ruolo di vettore di informazione genetica è, anche in questo caso, fornita.
I “mattoni” del DNA e dell’RNA
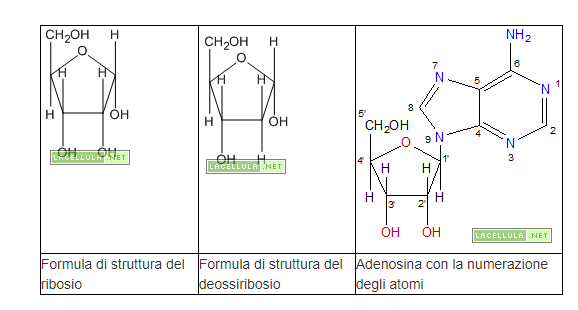
Quali sono i “mattoni”, ovvero gli elementi, che determinano la sequenza del DNA? Le strutture biomolecolari che troviamo lungo la doppia elica sono basi azotate puriniche o pirimidiniche legate ad uno zucchero deossiribosio, nel caso del DNA, o ribosio, nel caso dell’RNA. Sia il ribosio che il deossiribosio sono degli zuccheri chetonici a cinque atomi di carbonio uniti tra loro mediante dei ponti fosfodiesterici.
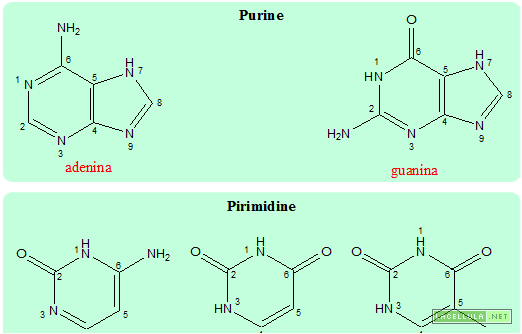
Le basi puriniche sono rappresentate dall’adenina e dalla guanina mentre le basi pirimidiniche sono la citosina, l’uracile (presente nell’RNA) e la timina (presente nel DNA).
Quando alle basi puriniche si associa lo zucchero (deossi-)ribosio allora si forma un nucleoside. Se volessimo indicare il numero per ogni atomo formante il nucleoside ci troveremmo di fronte ad un problema: da quale carbonio partire?
Il metodo standard è quello di intendere gli atomi delle basi azotate come “normali” o in altri termini atomi da cui partire ed identificarli con la lettere C (nel caso del carbonio) mentre i carboni dello zucchero (deossi-)ribosio come carboni “aggiunti” ed identificarli aggiungendo il suffisso ‘, che si pronincia “primo” accanto al numero del carbonio. Avremo così i carboni: C-1′, C-2′, C-3′, C-4′ e C-5’.
La disposizione tridimensionale delle basi nella doppia elica
Una disposizione senza un preciso ordine, infatti, non permetterebbe l’appaiamento delle basi che, come vedremo in seguito, risulta essere di fondamentale importanza per la costituzione della doppia elica. Schematicamente, come illustrato in figura, all’“interno” del DNA trovano posto le basi puriniche/pirimidiniche, mentre all’esterno gli zuccheri (deossi-)ribosio che sono legati da ponti fosfodiesterici in 3′ ed in 5′. Abbiamo appena illustrato cosa vuol dire l’apice nella nomenclatura per cui è facile capire che i ponti diesterici si legano ad un ossidrile in C’-3 ed in C’-5 dello zucchero (deossi-)ribosio.
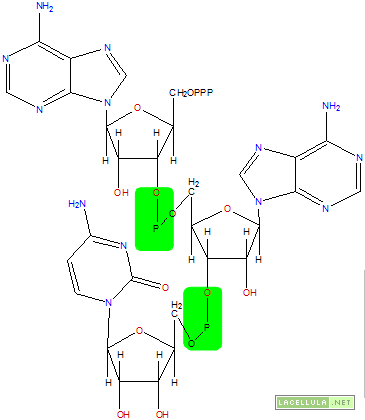
Supponiamo di avere la semplice sequenze AAC (adenosina-adenosina-citidina); la struttura sarà la seguente
Da come è possibile vedere l’ossidrile in 3′ lega l’ossidrile in 5′ di un ribosio che, a sua volta, è legato da un ossidrile in 3′.
La sequenza del DNA appena vista può essere ripetuta per decine, centinaia e migliaia di basi ed è permessa una certa flessione a livello dei legami fosfodiesterici. In questo modo è possibile che le basi di due catene opposte, ovvero di due catene antiparallele, possano trovarsi una dinanzi all’altra e instaurare dei legami idrogeno che sono molto deboli.
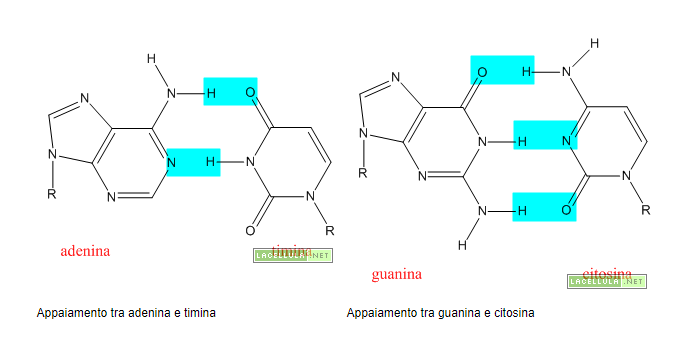
L’appaiamento di basi
| BASE | DNA | RNA |
| A | T | U |
| G | C | C |
| T | A | / |
| C | G | G |
| U | / | A |
I due filamenti di DNA, indifferentemente dalla loro lunghezza, sono incredibilmente resistenti. Per poterli separare o, utilizzando un sinonimo, denaturarli è necessario raggiungere temperature di oltre 90°C o agire sui parametri fisici della soluzione, ad esempio modificando il pH.
La resistenza del doppio filamento potrebbe indurre a pensare che i filamenti di DNA sono uniti da fortissimi legami. In realtà, i due filamenti antiparalleli, sono uniti da deboli legami idrogeno. Come si spiega l’elevata resistenza? Se consideriamo che ogni base di un filamento si appaia, contraendo legami deboli ad idrogeno, con un’altra base risulta chiaro che un elevato numero di legami idrogeno, proporzionale al numero di appaiamenti, conferisce una globale resistenza alla molecola di DNA. Gli appaiamenti, salvo in situazione patologiche o comunque straordinarie, seguono lo schema A-T, G-C per quello che riguarda il DNA e A-U G-C per quello che riguarda l’RNA.
Superavvolgimento del DNA
Abbiamo avuto modo di vedere come i due filamenti che costituiscono il DNA sono ordinatamente disposti a formare una doppia elica in quanto si intrecciano lungo un ipotetico asse.
Molti virus possiedono, al pari di alcuni organismi eucariotici, delle molecole di DNA circolari che, come il nome stesso suggerisce, non hanno delle terminazioni vere e proprie. In queste molecole si può assistere ad una ulteriore torsione della molecola circolare di DNA che forma il cosiddetto DNA superavvolto.
DNA rilassato, circolare e superavvolto
Linking number
Il linking number, abbreviato Lk, è un numero che definisce il livello di avvolgimento del DNA o, più precisamente, di qualsiasi struttura che possa essere avvolta. Una molecola di DNA rilassata ha un Lk0 che può essere determinato dividendo il numero di basi per il numero di basi per giro di elica. Se consideriamo il virus SV40, che ha un genoma di circa 5000 basi e 10.4 basi per giro di elica quando il DNA si trova in forma rilassata avremo che:
Lk0 = 5000 / 10.4 = 480 circa
Per calcolare il linking number di una molecola di DNA superavvolta si usa la semplice equazione:
Lk = Tw + Wr
dove
Tw = Twist, ovvero il numero di volte che un filamento di DNA incrocia un altro filamento. Da notare che Tw = LK0
Wr = Writhe, ovvero il numero di volte che, per ciò che concerne la biologia del DNA, la doppia elica si ripiega su sé stessa.
NA A, DNA B e DNA Z
| Nome | Passo | Basi | Verso | Conformazione |
| DNA B(comune) | 34Å | 10 | Destrorso | Anti |
| DNA A | 24Å | 11 | Destrorso | Anti |
| DNA Z | 46Å | 12 | Sinistrorso | Syn (purine) |
La configurazione più comune è quella definita DNA B che possiede un passo, ovvero la distanza tra un elica ed un’ altra, di 34Å ed un numero di basi per passo pari a 10. Inoltre l’andamento della molecola di DNA è di tipi destrorso. Il DNA A è relativamente poco presente in natura ed è caratterizzato da un passo pari a 24Å che contiene dieci basi ad andamento destrorso.
Un tipo di DNA, anche questo poco comune, è quello definito DNA Z che possiede andamento sinistrorso ed un passo di 45Å nel quale trovano posto 12 basi.
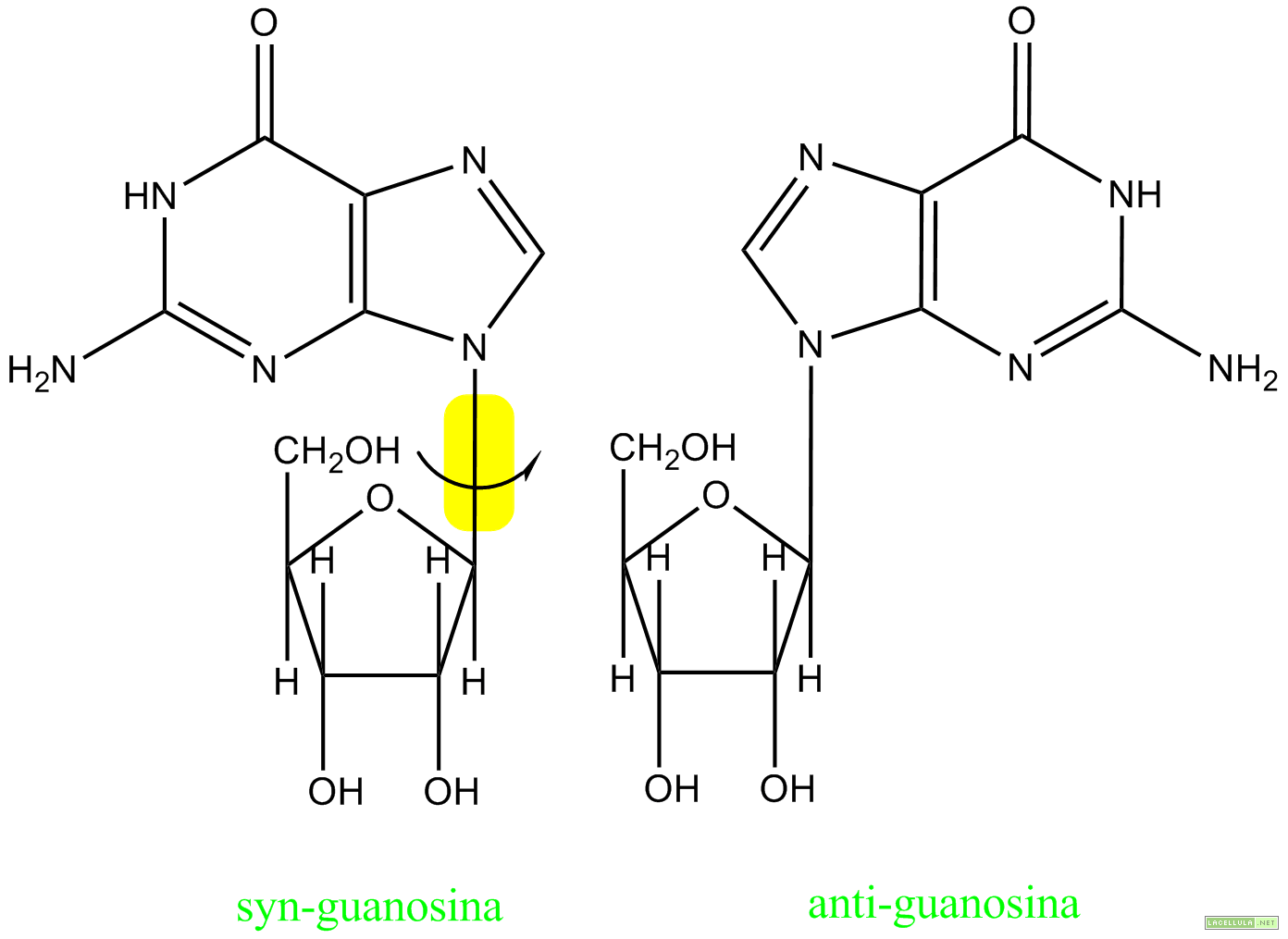
La differenza tra i tipi di DNA sembra essere dovuta a differenti gradi di idratazione della molecola, dalla presenza di determinati ioni che possono interagire con le basi o con gli zuccheri che costituiscono la doppia elica e dalla disposizione anti o syn degli elementi nucleotidici. Non è da sottovalutare anche l’interazione con particolari proteine, tra cui gli istoni che analizzeremo in dettaglio più avanti, che possono modificare la forma del DNA.