Indice
Gli acidi nucleici sono macromolecole polimeriche lineari ossia dei polimeri di nucleotidi i cui monomeri sono i nucleotidi stessi. Hanno grande importanza a livello biologico in quanto contengono e trasportano tutta l’informazione genetica di un organismo. Data la loro funzione si trovano principalmente all’interno del nucleo , la zona maggiormente protetta della cellula ma in misura minore anche nel citoplasma. Negli organismi viventi si trovano due tipi di acidi nucleici:
a) DNA (acido desossiribonucleico o deossiribonucleico)
b) RNA (acido ribonucleico)
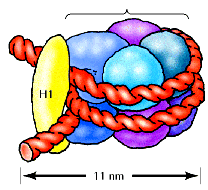
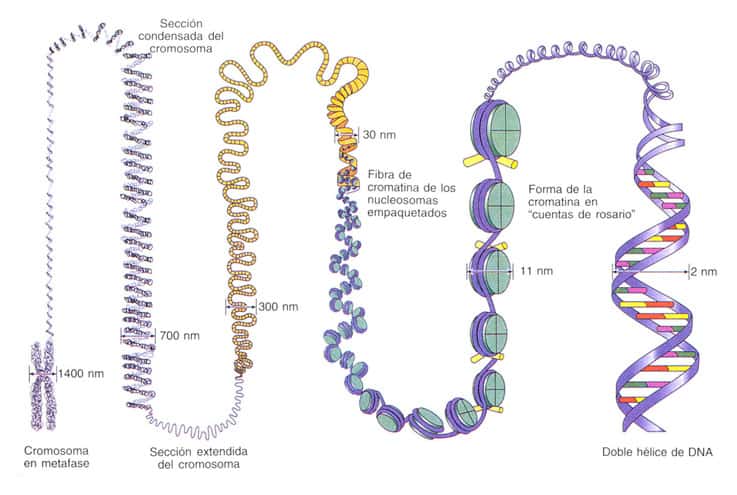
Nelle cellule procariote (dove non esiste il nucleo) tutte le informazioni sono contenute in una sola molecola di DNA circolare mentre nelle cellule eucariote il DNA si trova all’interno del nucleo in una forma detta cromatina, che consiste sostanzialmente di filamenti di DNA ripiegati in molti modi diversi attorno a delle proteine chiamate istoni (H1, H2A, H2B, H3, H4) , formando delle strutture piuttosto complicate, i cromosomi, caratteristici per forma e numero. Gli istoni possiedono un alto contenuto di catene laterali cariche positivamente e circa un residuo ogni quattro è rappresentato da lisina o da arginina. Ciascun tipo di istone può esistere in una varietà di forme a causa di modificazioni post-traduzionali. La modulazione della carica, della capacità di formare legami idrogeno e della forma degli istoni ad opera di queste modificazioni covalenti reversibili è molto importante per il compattamento del DNA e per la regolazione della sua disponibilità ad essere replicato e trascritto. Inoltre, le sequenze degli aminoacidi degli istoni H3 e H4 essendo quasi le stesse in tutti i vegetali e in tutti gli animali indicano che queste strutture così conservate sono in grado di svolgere un ruolo critico stabilito molto presto nell’evoluzione degli eucarioti e rimasto praticamente invariato nel tempo.
Ma in che modo gli istoni si combinano con il DNA per formare la fibra di cromatina?
Fu Arthur Kornberg a proporre nel 1974 che la cromatina fosse costituita da unità ripetitive , ciascuna contenente 200 coppie di basi di DNA e due molecole di ciascun istone H2A,H2B,H3 e H4. Queste unità ripetitive prendono il nome di nucleosomi.
La maggioranza del DNA è quindi avvolta intorno ad un nucleo centrale di istoni. La parte rimanente di DNA chiamata linker o DNA di congiunzione unisce nucleosomi adiacenti e contribuisce alla flessibilità della fibra di cromatina. Pertanto, una fibra di cromatina è una catena di nucleosomi uniti fra loro da giunzioni flessibili in maniera simile alle perle di una collana.
Tale modello di struttura cromatinica è in accordo con una serie di evidenze sperimentali :
a) Microscopia elettronica (osservazione di insiemi lineari di particelle sferiche di 100°A di diametro connesse da un sottile filamento)
b) Diffrazione ai raggi X e neutronica (indicazione che il DNA si trova all’interno del nucleosoma)
c) Digestione con nucleasi
d) Ricostituzione (si può formare una fibra simile a quella della cromatina in vitro aggiungendo istoni a DNA di virus. Studi di diffrazione ai raggi X hanno confermato che H2A,H2B, H3 e H4 devono essere aggiunti al DNA per ottenere il profilo di diffrazione della cromatina mentre H1 non è invece necessario e questo è in accordo con i dati che indicano che H1 non è parte integrante del nucleosoma).
Altro quesito : “in che modo il DNA viene compattato nei cromosomi ? ”
E’ stato Bruno Zimm a superare questo problema per mezzo di una tecnica viscosoelastica che forniva una misura delle dimensioni delle molecole più grandi di DNA in una miscela. Tale tecnica ha rivelato che un cromosoma di Drosophila M. contiene una molecola di DNA non interrotta. Inoltre, tale DNA è lineare e senza ramificazioni. Questa tecnica rende possibile analizzare e separare molecole di DNA molto più grandi di quanto non fosse possibile fare prima utilizzando un campo elettrico pulsante in un’unica direzione (pulsed field elcetrophoresis).
Gli istoni possono interagire con la maggior parte delle sequenze di DNA in accordo con il loro ruolo di componenti essenziali del meccanismo per la compattazione del DNA. I dimeri H2A-H2B sono attaccati a ciascuna delle due estremità esposte del tetramero H3-H4 per stabilizzare ulteriormente il nucleo del nucleo soma. L’istone H1 svolge invece un ruolo chiave al livello successivo di struttura del cromosoma servendo da ponte fra nucleosomi adiacenti. H1 si trova all’interno del nucleosoma vicino al DNA linker dove interagisce con H2A del nucleo e viene rilasciato dai nucleosomi quando il DNA delle particelle viene ulteriormente tagliato. Esso è anche diverso perché è presente in una unità per nucleosoma invece che in due come gli altri istoni.
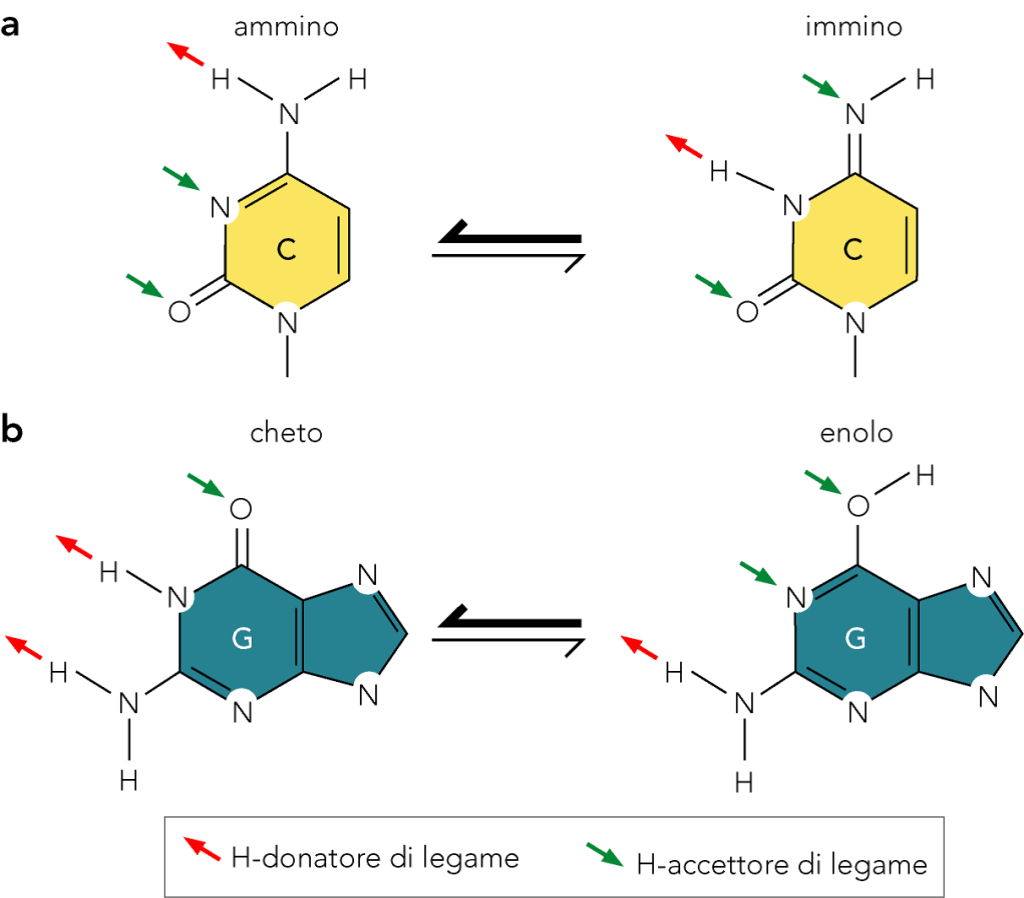
Inoltre, H1 è fosforilato prima della mitosi e poi defosforilato subito dopo. Questa modificazione covalente sembra essere coinvolta nella regolazione della sua capacità di compattare il DNA. La struttura del DNA in natura ed in condizioni normali viene detta del tipo B destrogira con le basi perpendicolari all’asse dell’elica; in condizioni particolari esistono altre due strutture dette A e Z. La prima struttura non esiste in vivo e corrisponde alla molecola disidratata. La seconda struttura è un elica sinistrorsa dove lo scheletro presenta un andamento ondulato , una percentuale di basi G e C è decisamente alta e ciò potrebbe essere un riconoscimento particolare.
2 molecole di H2A, H2B, H3 e H4
I livelli di super avvolgimento sono per il DNA delle cellule eucariote, una caratteristica fondamentale. Questo superavvolgimento si può pensare come un filo avvolto a formare una matassa e tante matasse legate assieme formano una struttura super avvolta. Nel minimo spazio sono contenute enormi informazioni genetiche. L’avvolgimento avviene grazie alle proteine presenti in associazioni con l’acido, l’unità elementare è detta nucleosoma ( unità strutturale della cromatina : rappresenta un ottamero di proteine istoniche [ due ciascuno di H2A,H2B,H3 e H4] su cui si avvolge il DNA). Sul nucleosoma si avvolgono 140 coppie di basi cioè quasi due giri completi di DNA) bloccati in posizione da una molecola di istone H1).
Il super avvolgimento prende il nome di polinucleosoma.
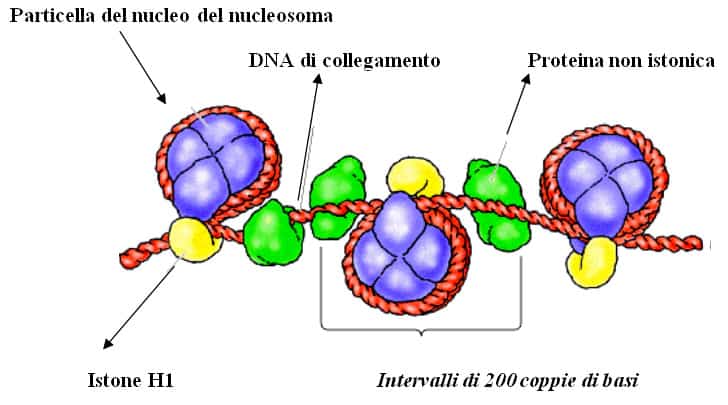
La cromatina contiene, inoltre, quantità approssimativamente analoghe di un’ampia varietà di proteine non istoniche. Vi sono più di un migliaio di differenti tipi di proteine, che sono coinvolte in varie attività funzionali, come la replicazione del DNA e l’espressione genica. Il compattamento del DNA con gli istoni produce una fibra di cromatina con un diametro di circa 10 nm che è composta da nucleosomi separati da segmenti di DNA di collegamento lunghi in media 80 coppie di basi. Il compattamento del DNA in queste fibre di cromatina accorcia la sua lunghezza di circa 6 volte. La cromatina può poi essere ulteriormente condensata da un avvolgimento in fibre di 30 nm la cui struttura rimane ancora da determinare. Le fibre di cromatina da 30 nm contengono circa 6 nucleosomi per giro.
I COMPONENTI DEGLI ACIDI NUCLEICI
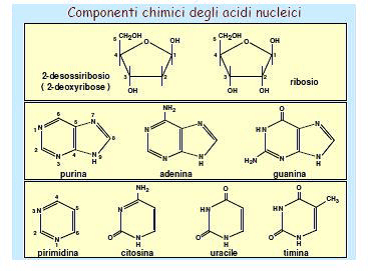
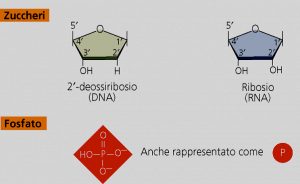
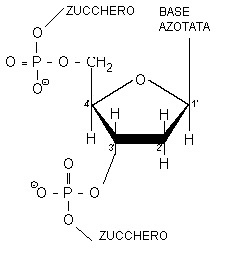
I nucleotidi oltre ad essere costituenti del DNA e dell’RNA sono composti ricchi di energia. Essi si trovano anche come trasduttori secondari di segnali chimici portati da alcuni ormoni e come componenti della struttura di importanti coenzimi. I nucleotidi sono formati da uno zucchero a 5 atomi di carbonio (un pentoso), da una base azotata e da gruppi fosforici.
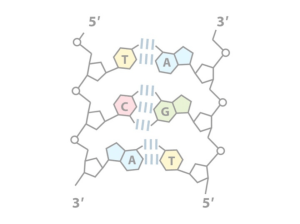
Il genoma umano è costituito da molecole di DNA che compongono i cromosomi, presenti nel nucleo e nei mitocondri. Il DNA è il biopolimero che contiene tutto il codice genetico di un organismo vivente L’uomo possiede (nel nucleo) 23 coppie di cromosomi, ognuno dei quali è composto da una molecola di DNA. La molecola di DNA è formata da due filamenti antiparalleli (denominati senso e antisenso) (orientati in direzione 5’-3’ e 3’-5’) che si avvolgono su loro stessi a formare una doppia elica unita da ponti idrogeno. Questa catena è larga poco più di 2 nanometri (nm) ma la lunghezza può arrivare a dimensioni dell’ordine del metro. Per essere contenuta in una cellula, che ha dimensioni di qualche micron (μm), deve, ripiegarsi attorno agli istoni, costituendo in tal modo la cromatina. I suoi monomeri costituenti come già riferito sono detti nucleotidi e precisamente deossiribonucleotidi.
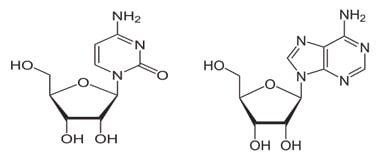
In entrambi i due acidi nucleici vi sono due tipi di basi azotate: le puriniche (anello doppio) : a) adenina e guanina e le pirimidiniche (anello semplice): b) timina, citosina e uracile. Le basi azotate che costituiscono il DNA sono adenina (A),guanina (G), citosina (C) e timina (T) mentre nell’RNA le basi azotate sono rappresentate da adenina (A), guanina(G), citosina(C) e uracile (U) al posto della timina.
Le basi azotate degli acidi nucleici
La doppia elica di DNA accoppia una pirimidina e una purina ( composti organici eterociclici): l’adenina si accoppia con la timina e la citosina con la guanina tramite legami idrogeno, che possono essere rotti con relativa semplicità. L’RNA (anche se singola catena) accoppia durante le trasmissioni e le traduzioni l’adenina all’uracile (la timina non è presente nell’RNA) e la citosina alla guanina. Gli atomi di carbonio sono numerati a partire da quello che si lega alla base. I legami tra gruppo fosfato e zucchero, e quindi tra i nucleotidi, si hanno ad opera degli atomi di Carbonio 3′ e 5′.
I nucleotidi di ogni filamento di DNA sono uniti tra di loro da un legame covalente che si forma tra l’acido fosforico di un nucleotide e lo zucchero del nucleotide successivo. I due filamenti sono a loro volta uniti da legami idrogeno (legami deboli) che si instaurano fra le basi azotate, una purina ed una pirimidina, in particolare: 1. Adenina – Timina: 2 legami idrogeno (deboli) 2. Guanina – Citosina: 3 legami idrogeno (deboli)
I due filamenti del DNA interagiscono fra loro attraverso una rete di legami a idrogeno che si stabiliscono rispettivamente tra citosina e guanina e tra adenina e timina.
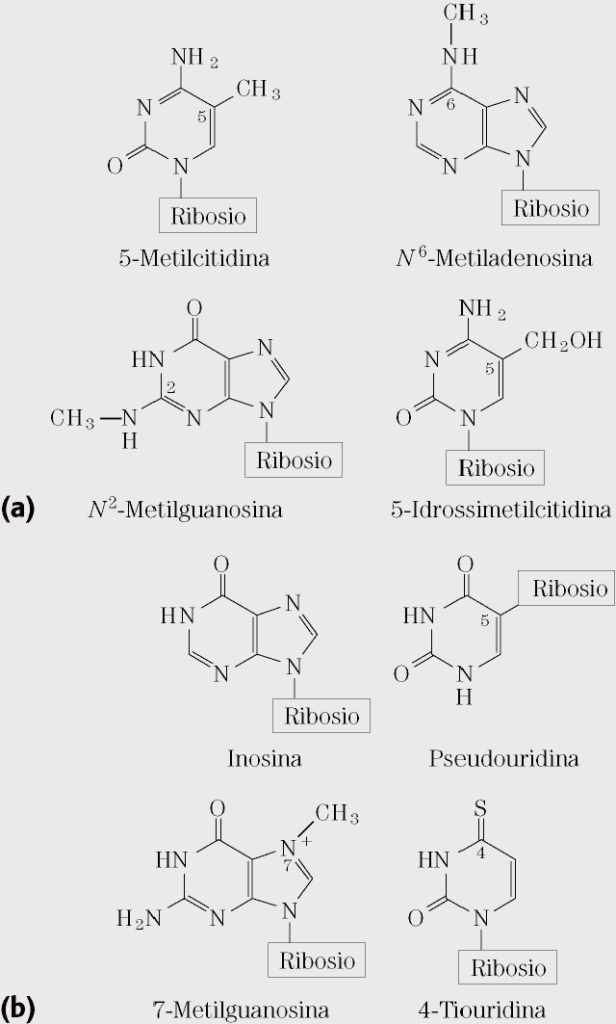
Altri importanti basi azotate derivate ad esempio dalla purina (il cui nucleo risulta dalla condensazione, in corrispondenza degli atomi 4 e 5, di un anello pirimidinico e di uno imidazolico) sono rappresentati da : ipoxantina (6-ossipurina), xantina (2,6-diossipurina) e l’acido urico (2,6,8-triossipurina) i quali sono i prodotti successivi del catabolismo degli acidi nucleici.
a) basi azotate minori del DNA b) basi azotate minori dei tRNA .
Nell’ RNA può essere anche presente l’inosina, base purinica non standard necessaria nella corretta traduzione di certi tipi di RNA virale.
I pentosi degli acidi nucleici possono ciclizzare per formare anelli furanosici
Il D- ribosio chimicamente è un monosaccaride che fa parte degli aldo-pentosi ( zucchero a 5 atomi di C) avente, in forma aperta , tutti i gruppi ossidrili o -OH rivolti verso destra. La forma aperta si converte in forma chiusa (prevalente in soluzione) mediante una reazione intramolecolare. Infatti, le forme predominanti in soluzione non sono le catene aperte di questi zuccheri perché queste catene tendono a ciclizzarsi generando degli anelli. Un’aldeide in genere reagisce con un gruppo alcolico per formare un emiacetale.
L’atomo di carbonio 1 (C-1) dell’aldeide nella forma a catena aperta del ribosio reagisce con il gruppo ossidrilico sull’atomo di carbonio C- 4, formando così un emiacetale intramolecolare. Quindi, come per tutti gli esosi anche per i pentosi che presentano un sufficiente numero di atomi per potersi chiudere ad anello, la forma aperta è in equilibrio con la forma ciclica emiacetalica.
Reazione di equilibrio e reversibile tra la forma aperta e quella emiacetalica del ribosio anche se normalmente l’equilibrio è più spostato verso la forma emiacetalica.
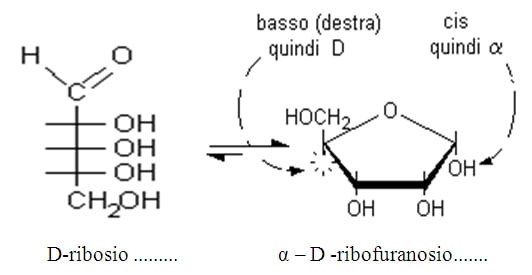
Poiché l’anello del ribosio è costituito da cinque atomi (quattro di carbonio e uno di ossigeno), si dice che il D – ribosio assume forma furanosica, principalmente β – furanosica. L’anello furanosico può esistere nelle configurazioni α e β ( la ciclizzazione può dar luogo a due diverse strutture chiamate anomeri in quanto differiscono solo per la posizione del gruppo OH legata al C-1 rispetto al piano dell’anello) a seconda che il nuovo gruppo -OH, ottenuto dall’apertura del doppio legame carbonilico, giaccia al di sotto o al di sopra del piano medio della molecola. Il C-1 che nella forma emiacetalica diventa chirale è detto C asimmetrico . Il D- ribosio ciclico è presente in forma trans (α) o in forma cis (β). La forma anomerica β è la più diffusa e presenta il gruppo OH legato al carbonio 1 in posizione equatoriale..
Gli anelli emiacetalici a sei termini vengono indicati dall’infisso –piran mentre quelli a cinque dall’infisso –furan in analogia ai nomi dei composti pirano e furano.
I simboli D o L (enantiomeri) si riferiscono alla configurazione assoluta del Carbonio asimmetrico più lontano dal gruppo carbonilico. Nel caso dei due pentosi , il simbolo D distingue la posizione dell’OH sul penultimo carbonio della catena, il C-4) Se la molecola ha questo OH rivolto a destra nelle proiezioni di Fischer viene denominata D mentre se è a sinistra viene denominata L.
L’anello ribofuranosico può esistere in altre diverse conformazioni
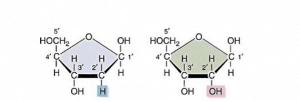
L’anello furanosico del ribosio non esiste come struttura perfettamente planare. Non è un pentagono come lo si usa disegnare secondo le proiezioni di Haworth (struttura emiacetalica) ma possiede una struttura tridimensionale in cui i quattro elementi dell’anello (i vertici del pentagono) giacciono sullo stesso piano e un quinto membro dell’anello fuori dal piano. Questa conformazione viene chiamata a busta in quanto la sua struttura ricorda riguarda la forma di una busta aperta con la lingua sollevata.
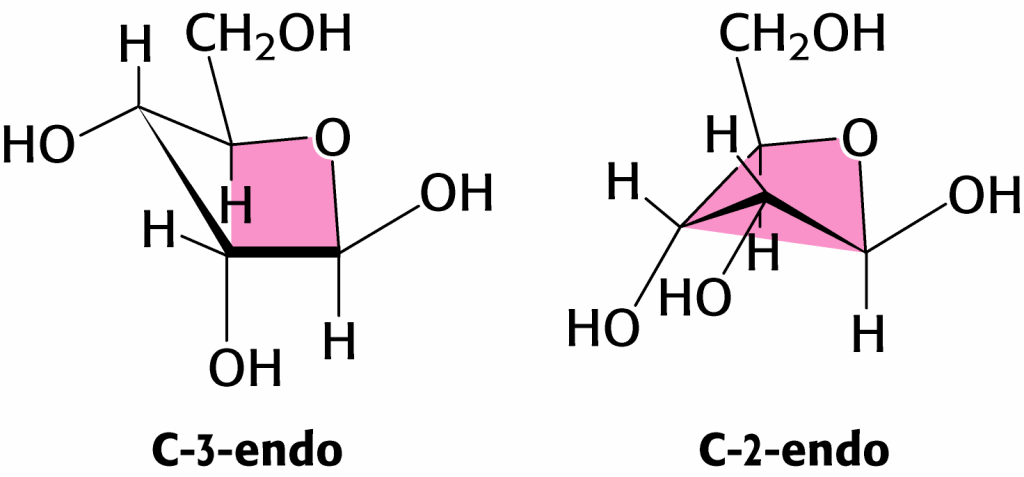
Nell’unità di ribosio della maggior parte delle biomolecole in genere sono gli atomi di carbonio C-2 o C-3 ad assumere questa conformazione essendo fuori del piano cioè sullo stesso lato del piano dove si trova l’atomo di carbonio C-5 ( configurazione C- endo) oppure sul lato opposto dell’atomo C-5 (conformazione C-eso) mentre C-1, C-2, C- 4 e l’atomo di ossigeno O giacciono sullo stesso piano ( sono coplanari) . Pertanto, la presenza di un gruppo OH in posizione 2′ del ribosio presente nella molecola di RNA forza il ribosio nella conformazione C3′-endo, a differenza della C2′-endo tipica del deossiribosio, generando così due molecole comunque differenti tra loro. Anche la nomenclatura sin/anti viene spesso usata.
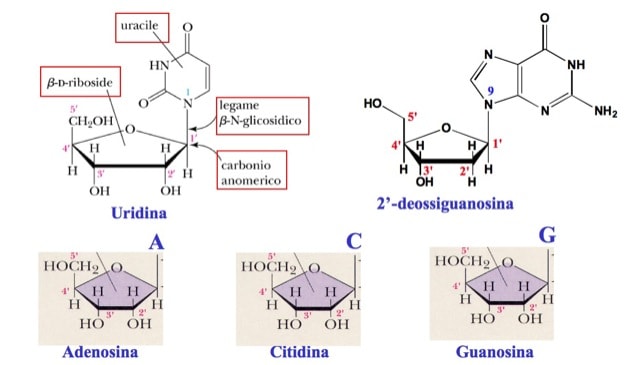
Il D- ribosio si lega ad una base azotata attraverso legame β-glicosidico ( legame β- N1 –glicosidico nei nucleosidi pirimidinici e legame β- N9 – glicosidico nei nucleosidi purinici) , che si forma per perdita di una molecola di acqua tra il carbonio 1 e il gruppo amminico libero della base azotata (si forma così un nucleoside, che avrà nomi diversi a seconda della base azotata legata). Il D-ribosio può inoltre formare un legame fosfodiesterico con l’ossigeno di un residuo di acido fosforico e il suo carbonio 5 o 3. In questo caso si parla di nucleotide, composto costituito da una base azotata, un residuo di D -ribosio e da un gruppo fosfato.
La conformazione della catena zuccheri fosfati
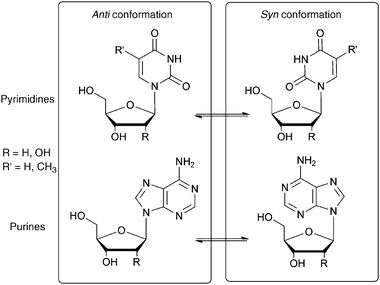
La conformazione di un’entità nucleotidica è specificata da sei angoli di torsione dello scheletro di zuccheri- fosfati e dall’angolo di torsione che descrive l’orientamento della base intorno al legame glicosidico (il legame che unisce C’1 alla base). Potrebbe sembrare che questi sette gradi di libertà per nucleotide possano rendere i polinucleotidici altamente flessibili. Ma in realtà, questi angoli di torsione sono soggetti ad una varietà di limitazioni interne che restringono enormemente la loro libertà conformazionale. Ad esempio, la rotazione di una base intorno al suo legame glicosidico è enormemente ostacolata. I residui di purine hanno due orientamenti stericamente permessi relativamente allo zucchero, noti come la conformazione syn (dalla stessa parte) e anti (parte opposta). Per le pirimidine, si forma facilmente solo la conformazione anti perché nella conformazione syn il residuo di zucchero interferisce stericamente con il sostituente C’2 della pirimidina.
Comunque, nella maggior parte degli acidi nucleici a doppia elica tutte le basi sono nella conformazione anti. L’eccezione è il DNA-Z, i cui residui alternati di pirimidine e purine sono rispettivamente anti e syn. Anche l’anello di ribosio ha un certo grado di flessibilità che influenza significativamente la conformazione dello scheletro di zucchero fosfato. Gli angoli al vertice di un pentagono regolare sono di 108°, un valore molto vicino all’angolo tetraedrico (109,5°), così che ci si potrebbe aspettare che l’anello di ribofuranosio sia quasi piatto. Tuttavia, i sostituenti dell’anello sono eclissati quando l’anello è planare. Per alleviare l’affollamento che ne risulta, che si riscontra anche tra gli atomi di idrogeno, l’anello si piega. Esso cioè diventa leggermente non planare in modo da riorientare i sostituenti dell’anello. In conclusione risulta che quattro atomi dell’anello risultano coplanari mentre il restante atomo è fuori da questo piano, una conformazione detta a mezza sedia. Se l’atomo di fuori del piano è spostato dallo stesso lato dell’anello rispetto all’atomo C’5 si dice che ha la conformazione endo, mentre se è spostato nel lato opposto dell’anello rispetto a C’5 ha la conformazione eso. Nella maggior parte delle strutture note di nucleosidi e nucleotidi l’atomo fuori dal piano è o C’2 o C’3 . C’2-endo è il ripiegamento del ribosio che si trova più frequentemente, assieme anche a C’ 3-endo e -eso.
DAI NUCEOSIDI AI NUCLEOTIDI
Nucleosidi
La reazione di condensazione di una di una delle basi azotate che costituiscono gli acidi nucleici con il ribosio o il desossiribosio forma un nucleoside.

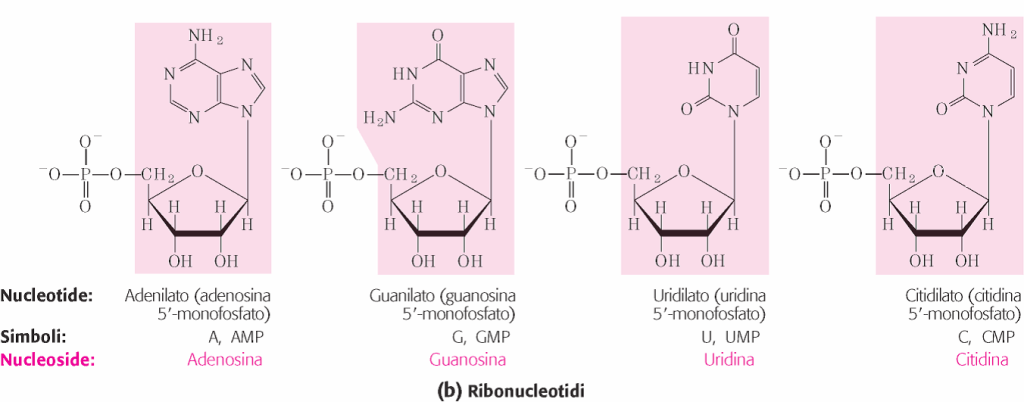
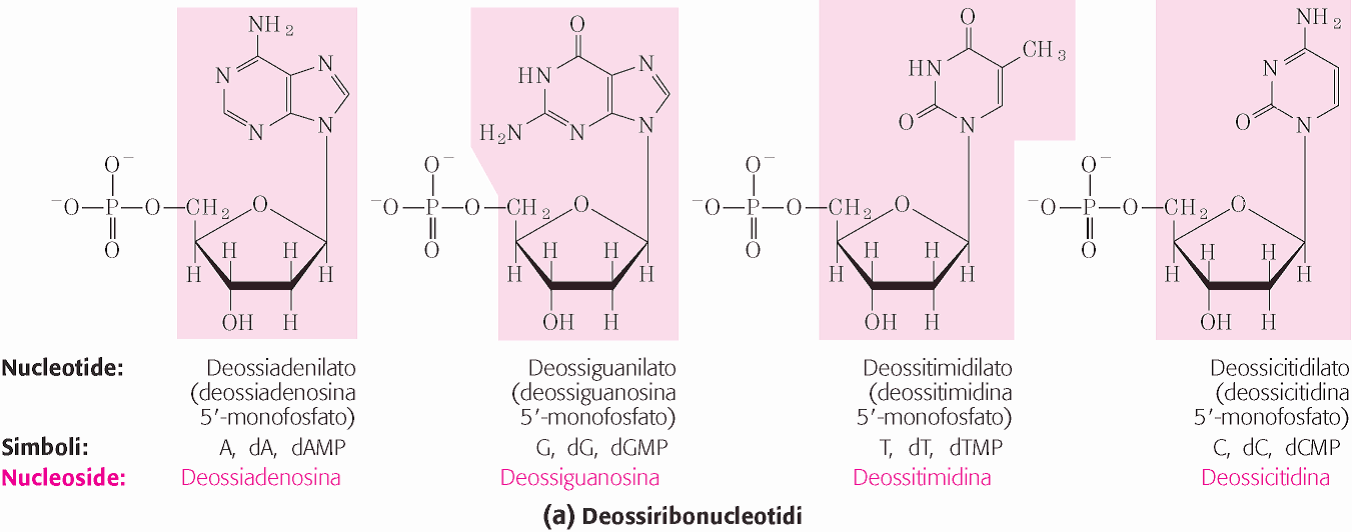
Un nucleoside consiste quindi di una base purinica e pirimidinica legata ad uno zucchero. I quattro nucleosidi del DNA si chiamano deossiadenosina, deossiguanosina, deossitimidina e deossicitidina mentre i nucleosidi dell’RNA prendono rispettivamente il nome di adenosina, guanosina, citidina e uridina.
Nel nucleoside dell’RNA , il Carbonio C-1 del ribosio è legato all’Azoto N-1 di una pirimidina o all’N-9 di una purina. La configurazione di questo legame N- glucosidico è β (in quanto la base si trova sopra il piano dell’anello dello zucchero).
In un deossiribonucleoside, il Carbonio C-1 del deossiribosio è legato all’N-1 di una pirimidina o all’N-9 di una purina. La configurazione di questo legame N-glicosidico è β ( la base si trova sopra il piano dell’anello dello zucchero) e lo stesso vale per i ribonucleosidi.
Nucleotidi
Un nucleotide è costituito da tre unità strutturali condensate tra loro: una delle 5 nucleobasi, uno zucchero e una o più molecole di acido fosforico.
I deossinucleosidi, per aggiunta di un gruppo fosfato, formano i deossinucleotidi, i monomeri del DNA, mentre i ribonucleosidi dell’RNA.formano i ribonucleotidi
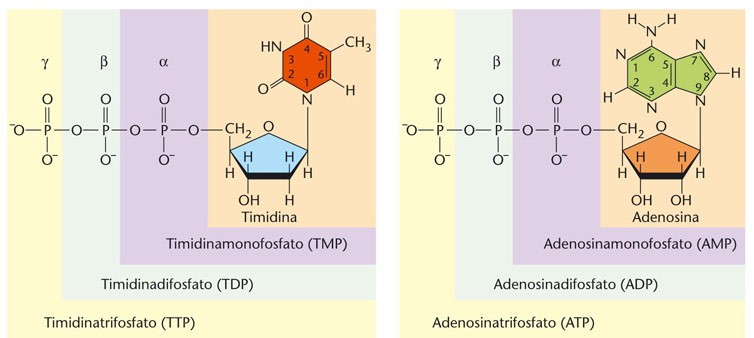
Un nucleotide prende il nome dal nucleoside che contiene. Per es. l’ adenosina monofosfato (dAMP) è il nucleotide che contiene adenosina e un fosfato. Un legame estereo che coinvolge il fosfato è chiamato legame fosfoestereo.
Il nucleotide è quindi l’estere fosforico di un nucleoside. Il sito più comune di esterificazione nei nucleotidi naturali è il gruppo ossidrile -OH attaccato al carbonio C-5 dello zucchero. Questo composto prende il nome di nucleoside 5’ fosfato oppure 5’- nucleotide. Ad es. la deossiadenosina 5’ trifosfato (dATP) è un precursore attivato nella sintesi del DNA.
Un nucleotide è attivato dalla presenza di due legami fosfoanidrilici presenti nelle unità trifosfato. L’aggiunta di uno , due o tre residui fosforici nella catena produce rispettivamente i nucleosidi mono, difosfato e trifosfato (NMP, NDP e NTP) che sono fondamentali nel metabolismo energetico della cellula. I più importanti sono ADP e ATP, rispettivamente adenosina difosfato e adenosina trifosfato. Il passaggio da tri- a di- avviene tramite una reazione di idrolisi esoergonica con conseguente liberazione di energia che la cellula può utilizzare per le sue attività. Ad es. nel caso della deossiadenosina 5’ trifosfato o dATP che è un precursore attivato del DNA il prefisso “d” nel dATP indica che lo zucchero è il deossiribosio per distinguere questo composto dall’ATP in cui lo zucchero è invece il ribosio.
L’ossatura del DNA che è sempre uguale in tutta la molecola consiste di deossiribosi legati a gruppi fosforici . Esattamente l’OH 3’ dello zucchero di un deossiribonucleotide è unito all’OH 5’ dello zucchero adiacente da un ponte fosfodiesterico. La parte variabile del DNA è la sua sequenza di 4 specie di basi (A,G,C,T) . A causa della presenza del residuo fosforico conferente carattere fortemente acido, ai nucleotidi corrispondenti si da il nome il nome di acido desossiadenilico o deossiadenilato, acido deossiguanilico o deossiguanilato, acido deossicitidilico o deossicitidilato e acido desossitimidilico o deossitimidilato.
Funzioni dei nucleotidi
Molecole di riserva : l’idrolisi dei nucleosidi trifosfato costituisce una fonte di energia chimica per guidare un gran numero di reazioni biochimiche
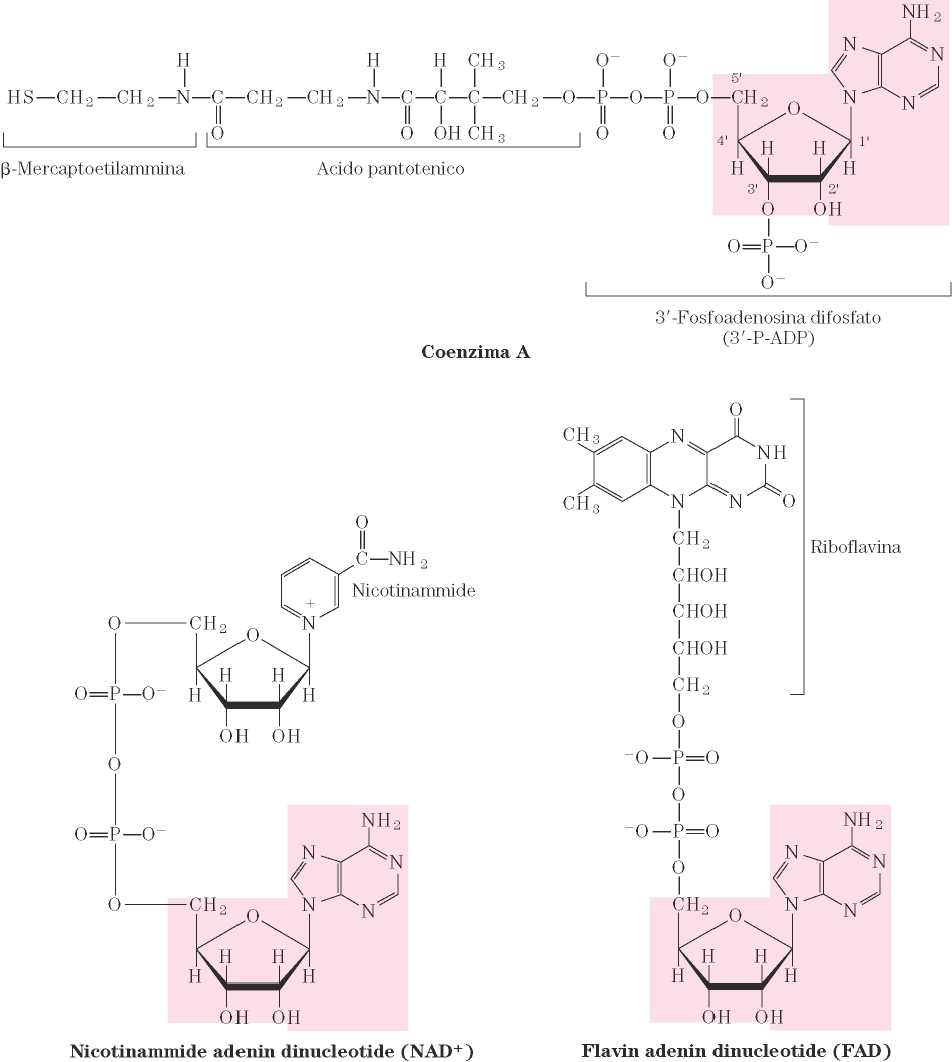
Precursori di coenzimi : es. CoA, NAD e FAD
Molecole regolatrici : es. cAMP e cGMP come secondi messaggeri oppure ppGpp che viene prodotto nei batteri in risposta ad una diminuzione della sintesi proteica che si ha durante una carenza di amminoacidi. Inibisce la sintesi di rRNA e tRNA non necessari in condizioni di bassi livelli di sintesi proteica.
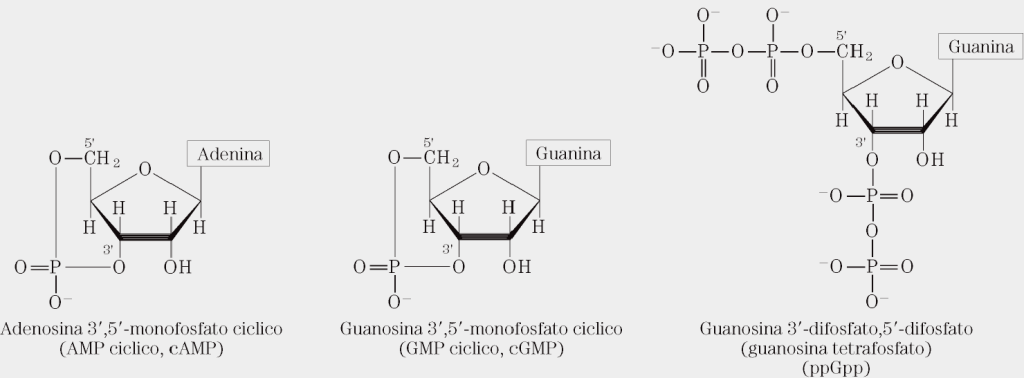
Mattoni da costruzione degli acidi nucleici